2018.11.13
「遺伝子のスイッチ役を「見える化」-バイオビッグデータを有効活用-」(発生再生医学分野 沖真弥助教・目野主税教授)
遺伝子のスイッチ役を「見える化」-バイオビッグデータを有効活用-
九州大学大学院医学研究院の沖 真弥助教と目野 主税教授は、情報システム研究機構・ライフサイエンス統合データベースセンター(DBCLS)の大田達郎特任研究員などとの共同研究により、世界中から報告されたタンパク質とゲノムDNAの結合情報を全てデータベース化し、組織や臓器を形成する司令塔的なタンパク質の探索に応用できることを示しました。
われわれの体のゲノム DNA にはヒストンや転写因子と呼ばれるタンパク質が結合し、遺伝子のオン・オフのスイッチ役としてはたらき、臓器の形成や疾患などに関与します。そのようなタンパク質がゲノム全体のどこに結合しているかを調べるため、2007 年に ChIP-seq 法という手法が開発され、これまで 10 万件近くもの実験結果が報告されています。しかしそのデータ量があまりに膨大なため、そのビッグデータから必要な知識を取り出すことは非常に困難でした。
本研究グループは、論文などで報告された ChIP-seq データを全て収集してスーパーコンピューターで統合解析し、それを「見える化」した Web サービス(ChIP-Atlas; https://chip-atlas.org)を開発しました。これによりどの転写因子が、どの細胞で、どの遺伝子に結合するかが容易に理解できるため、すでに遺伝子制御のしくみ、薬理作用、老化や疾患などの研究に世界中で利用されています。また同研究グループは、このビッグデータをさらに高次解析し、免疫細胞、肝臓、血管などを形成する司令塔的な転写因子の予測に応用できることを示し、将来的には再生医療などに応用できると期待されます。本研究成果は、JST ライフサイエンスデータベース統合化推進事業、日本学術振興会科学研究費、AMED 老化メカニズムの解明・制御プロジェクトの支援を受けており、2018 年11月 9 日午後8 時(日本時間)に、国際学術雑誌「EMBO Reports」オンライン版に掲載されました。
われわれの体のゲノム DNA にはヒストンや転写因子と呼ばれるタンパク質が結合し、遺伝子のオン・オフのスイッチ役としてはたらき、臓器の形成や疾患などに関与します。そのようなタンパク質がゲノム全体のどこに結合しているかを調べるため、2007 年に ChIP-seq 法という手法が開発され、これまで 10 万件近くもの実験結果が報告されています。しかしそのデータ量があまりに膨大なため、そのビッグデータから必要な知識を取り出すことは非常に困難でした。
本研究グループは、論文などで報告された ChIP-seq データを全て収集してスーパーコンピューターで統合解析し、それを「見える化」した Web サービス(ChIP-Atlas; https://chip-atlas.org)を開発しました。これによりどの転写因子が、どの細胞で、どの遺伝子に結合するかが容易に理解できるため、すでに遺伝子制御のしくみ、薬理作用、老化や疾患などの研究に世界中で利用されています。また同研究グループは、このビッグデータをさらに高次解析し、免疫細胞、肝臓、血管などを形成する司令塔的な転写因子の予測に応用できることを示し、将来的には再生医療などに応用できると期待されます。本研究成果は、JST ライフサイエンスデータベース統合化推進事業、日本学術振興会科学研究費、AMED 老化メカニズムの解明・制御プロジェクトの支援を受けており、2018 年11月 9 日午後8 時(日本時間)に、国際学術雑誌「EMBO Reports」オンライン版に掲載されました。
 |
 |
【お問い合わせ】 | 九州大学大学院 医学研究院 発生再生医学分野 |
| 助教 沖 真弥 soki(a)dev.med.kyushu-u.ac.jp 電話:092-642-6259 教授 目野主税 meno(a)dev.med.kyushu-u.ac.jp FAX:092-642-6260 ※@を@に置きかえてメールを送信してください。 |
||
わたしたちの細胞の中に存在するゲノム DNA は、ヒストンと呼ばれるタンパク質に巻きついてクロマチンという構造を形成します。また、転写因子と呼ばれる約千種類のタンパク質がクロマチンに結合することで、付近の遺伝子発現のオン・オフを制御します。このような遺伝子のスイッチ役の巧みな制御によって、さまざまな組織や臓器などが形成され、正常に機能するようになります。もし転写因子に異常があると、遺伝子の制御が乱れるため、胎児の奇形、がんや自己免疫疾患などの原因となることが知られています。また、いくつかの転写因子は多数の遺伝子をまとめて制御できるため、そのような転写因子を皮膚の繊維芽細胞へ大量に導入すると、iPS 細胞や肝細胞などへ変えることもできます。
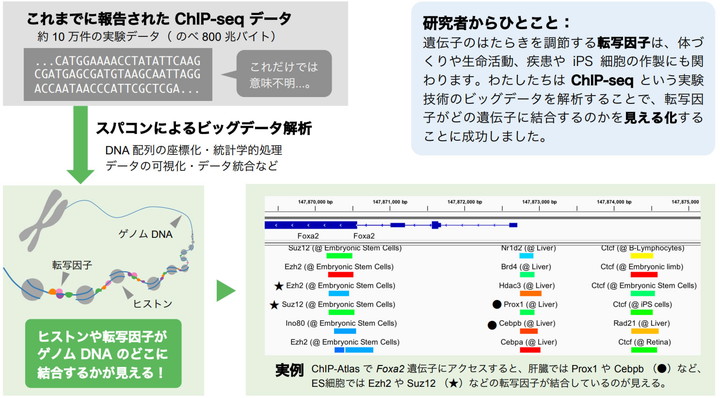
このように、転写因子は遺伝子のスイッチ役として機能しますが、30 億塩基対もあるゲノム DNA のどこに結合しているのか?については長らく謎のままでした。ところが約十年前に ChIP-seq※という手法が登場し、ヒストンや転写因子が結合する場所をゲノム全体にわたって調べることができるようになったため、遺伝子研究は大きなブレークスルーを迎えました。その後、ChIP-seq 法を用いた研究成果は世界中から次々に報告され、現在までに約 10 万件、800 兆バイトものデータ量になります。しかしこれらは A/T/G/C からなる DNA の塩基配列しか公開されていないため、そのデータの意味を理解して活用するためには複雑な計算処理やデータの可視化などが必要になります。この膨大な量のデータを計算するには、普通のパソコンをはるかに超えた能力の計算機が必要なため、これまで論文で報告されたChIP-seq データのほとんどは、他の研究者に活用されることなく死蔵されていました。
| ※ChIP-seq とは Chromatin immunoprecipitation with sequencing の略で、対象となるヒストンや転写因子に対する抗体を用いることで、それらが結合する DNA を集め、高速シーケンサーで配列を読み取る、という手法です。ただし、その DNA 配列だけでは何も理解できないため、配列データの座標化、統計解析や可視化処理などが必要です。 |
■発表内容
●ChIP-seq データベースの開発
九州大学と情報システム研究機構・ライフサイエンス統合データベースセンター(DBCLS)を中心とする研究グループは、国立遺伝学研究所のスーパーコンピューターシステムを利用して約 10 万件のChIP-seq ビッグデータを全て収集・計算し、ヒストンや転写因子がゲノム DNA に結合する位置情報をすべて可視化することに成功しました。また、実験に用いられた試料に関する情報を整理し、全てのデータについて実験対象の細胞や転写因子の名前をきちんと明記しました。これらのデータは 2015 年 12月より ChIP-Atlas(https://chip-atlas.org)という Web サービスとして公開しましたが、その後、更新や改訂を重ね、このたび正式に論文として報告しました。この中では本サービスの内容と使い方、可視化処理の方法や、利用事例(後述)について述べられています。
ChIP-Atlas は、高校で習う程度の転写因子やゲノムに関する基礎知識があれば誰でも使うことができます。アトラス(地図帳)をめくるように好きな遺伝子にアクセスし、そこに結合する転写因子やヒストンの種類、および細胞タイプなどを視覚的に理解できます。たとえば1ページ目の図は、マウスのFoxa2 という、肝臓形成に重要な遺伝子にアクセスした例です。図の最上段にはFoxa2 遺伝子の部分(いわゆるエクソン)が青いバーとして示されています。図の中央には Foxa2 遺伝子の端の部分(いわゆる転写開始点)がありますが、そこには●印で示すように Prox1 や Cebpb などの転写活性化因子が liver(肝臓)で結合するということがわかります。また Foxa2 遺伝子の左側の部分には★印で示すようにEzh2 や Suz12 という転写抑制化因子が embryonic stem cells(ES 細胞)で結合していることがわかります。つまり肝臓や ES 細胞において、Foxa2 遺伝子はこのような転写因子によってオンまたはオフの制御を受けることがビジュアルで理解できます。このような転写因子などの結合部位が ChIP-Atlas には約8億か所も収録されており、このようなデータベースは世界初です。
●ChIP-seq データベースの応用事例
ChIP-seq データは世界中から毎月約 1,500 件も報告されていますが、これらのデータは毎月ChIP-Atlas に追加されているため、利用者は常に最新のデータを閲覧できます。毎月、国内外から約 10万ページビューのアクセスがあり、これまで約 50 報の論文に引用されています。おもに遺伝子発現を制御するしくみに関する研究のほか、薬の作用、がん、老化、生物進化などにかかわる転写因子の研究に応用されています。これらの利用事例は今回の論文にも述べられており、今後も幅広い生命科学研究の発展に寄与できると思われます。
また同研究グループは、組織特異的に発現する複数の遺伝子がどの転写因子によって制御されているか?について解析しました。たとえば、肝臓では発現するが、その他の臓器ではほとんど発現しないような遺伝子を「肝臓特異的遺伝子」としてリストアップし、それらの多くに共通して結合するような転写因子を ChIP-Atlas のビッグデータを用いて探索しました。その結果、HNF4A や FOXA2 などの転写因子が肝臓特異的遺伝子の多くに結合することがわかりました。つまり HNF4A や FOXA2 は肝臓で重要な複数の遺伝子の発現をまとめて制御する、司令塔的な転写因子であるといえます。実際に皮膚の線維芽細胞にこれらの転写因子を大量に導入すると、肝細胞へと分化転換できることがすでに証明されています(いわゆるダイレクト・リプログラミング技術)。また同研究グループは、血管内皮細胞特異的遺伝子には TAL1, FOS, JUN など、マクロファージ特異的遺伝子群には STAT1 や SPI1 などの転写因子が高頻度に結合することを示しました。これらの成果は、それぞれの細胞タイプの司令塔的な転写因子であると考えられるため、将来的にはダイレクト・リプログラミング技術による組織再生への応用が期待されます。
■発表論文
| 著者: | Shinya Oki, Tazro Ohta, Go Shioi, Hideki Hatanaka, Osamu Ogasawara, Yoshihiro Okuda, Hideya Kawaji, Ryo Nakaki, Jun Sese, and Chikara Meno |
| タイトル: | ChIP-Atlas: a data-mining suite powered by full integration of public ChIP-seq data. |
| 掲載誌: | EMBO Reports, 2018, e46255.(doi: 10.15252/embr.201846255) |
■謝辞
本研究は以下の研究費の支援により行われました。
・科学技術振興機構(JST)ライフサイエンスデータベース統合化推進事業(統合化推進プログラム)
・日本学術振興会科学研究費(25840087, 18KT0024, 16H06530, 26291051, 17H01571, 15K14529)
・日本医療研究開発機構(AMED)老化メカニズムの解明・制御プロジェクト(JP17gm5010003)

